研究人员实现RNA定点高效乙酰化修饰
来源:分子细胞科学卓越创新中心 2024-04-24 15:25
该研究重组真核生物tRNA乙酰化修饰活力,提出乙酰化修饰酶复合物Kre33-Tan1识别底物的“分子标尺”模型,实现tRNA与小RNA分子上的定点高效乙酰化修饰。
4月13日,中国科学院分子细胞科学卓越创新中心研究员周小龙团队,在《核酸研究》(Nucleic Acids Research)上以Activity reconstitution of Kre33 and Tan1 reveals a molecular ruler mechanism in eukaryotic tRNA acetylation为题发表研究论文。该研究成功重组真核生物细胞质tRNA乙酰化修饰活力,揭示乙酰化修饰酶复合物Kre33-Tan1对底物的专一性识别机制,实现tRNA及小RNA分子上的定点高效乙酰化修饰。
在细胞内所有RNA分子中,tRNA含有最为密集且种类最为多样的转录后修饰。目前,主要发现2种RNA乙酰化修饰种类,即N4-乙酰胞苷(ac4C)及N6-乙酰腺苷(ac6A)。ac4C在三界生物以及病毒来源的多种RNA中广泛存在,而ac6A只存在于古细菌来源的tRNA中。20世纪60年代,ac4C首先在真核生物的tRNASer、tRNALeu以及细菌延伸tRNAMet 【tRNAMet(e)】中被鉴定。
在细菌tRNA中,ac4C特异性地存在于tRNAMet(e)反密码子第一位(第34位,ac4C34)。ac4C34活力重组、结构解析以及遗传学数据表明,TmcA以及TmcAL蛋白质通过两种不同的机制,分别利用乙酰辅酶A或乙酸作为乙酰供体,催化tRNAMet(e) ac4C34的生物合成。ac4C34可以阻止tRNAMet(e)的CAU反密码子与异亮氨酸AUA密码子的配对,防止AUA密码子被解码为甲硫氨酸,从而确保了细菌蛋白质合成的精确性。就真核生物tRNA而言,ac4C专一性地存在于第12位(ac4C12)。虽然发现于上世纪60年代,直到2004年,Tan1被发现参与ac4C12形成,但是Tan1没有催化活性中心,被认为是一个RNA结合蛋白质。2015年,真核生物tRNA ac4C12修饰酶Kre33才被鉴定。Kre33主要定位于核仁,是rRNA前体加工成熟的辅助因子。近年来,人THUMPD1(Tan1同源蛋白质)系列点突变导致出生缺陷型神经发育异常。长期以来,真核生物tRNA ac4C12修饰从未被成功重组,导致ac4C12生物合成的分子基础、底物识别机制、相关疾病的分子机制等完全未知,也无法基于修饰机器在特定RNA分子上实现定点乙酰化修饰。
该研究通过分析核糖体亚基前体剪接复合体中的Kre33结构,结合AlphaFold预测的Tan1结构,合理设计表达质粒,获得高纯度的Kre33与Tan1蛋白质;通过条件优化,利用乙酰辅酶A作为乙酰化供体,实现tRNASer以及tRNALeu上的高效乙酰化修饰;系统研究Kre33-Tan1识别tRNA底物的识别机制,提出真核生物tRNA乙酰化修饰的“分子标尺”模型;在特定tRNA底物上实现100%乙酰化修饰效率,实现多种非乙酰化tRNA底物变体的高效乙酰化修饰;基于“分子标尺”模型,实现小RNA的位点特异性高效乙酰化修饰;通过制备乙酰化修饰的tRNASer以及tRNALeu,揭示乙酰化修饰对于tRNA的热动力学以及氨基酰化水平没有显著影响,可能在翻译延伸的转位过程中介导了tRNA与核糖体的相互作用。
该研究重组真核生物tRNA乙酰化修饰活力,提出乙酰化修饰酶复合物Kre33-Tan1识别底物的“分子标尺”模型,实现tRNA与小RNA分子上的定点高效乙酰化修饰。研究加深了对真核生物tRNA乙酰化修饰的分子机制的认识,开发了特定RNA分子定点乙酰化修饰工具,有助于进一步研究RNA乙酰化修饰的机制与生物学功能,包括鉴定潜在的乙酰化修饰的“阅读器”和“擦除器”。
研究工作得到科学技术部、国家自然科学基金委员会、中国科学院、上海市科学技术委员会的支持。
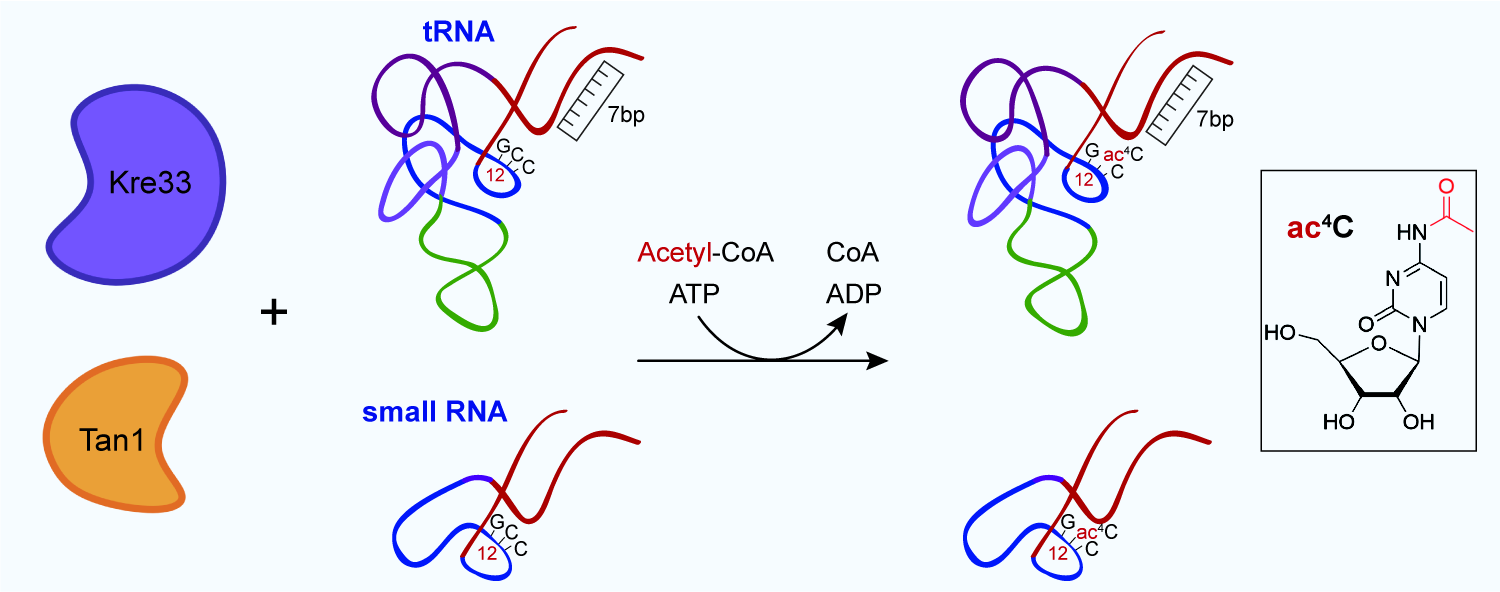
真核生物tRNA乙酰化修饰的实现与机制
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


